De conformidad con lo dispuesto en el Reglamento (UE) 2016/679 relativo a la protección de los datos personales de las personas físicas (RGPD), le informamos que la información personal que facilite quedará debidamente registrada e incorporada a los sistemas de tratamiento de datos responsabilidad de:
Denominación Social: Euroespes S.A.
Nombre Comercial: EuroEspes
Domicilio Social: Santa Marta de Babio S/N – Bergondo (A Coruña)
CIF / NIF: A15319452
Teléfono: 981780505
Fax: 981780511
e-Mail: info@euroespes.com
Inscrita en el Registro: R.M. de La Coruña, Tomo 855, Sección General, Folio 202, Hoja C-1635, Inscripción 1ª
Nombre de dominio: euroespes.com
Datos de contacto del Delegado de Protección de Datos: delegado@euroespes.com
Qué es la epigenética
Conecta los factores genómicos y los medioambientales que afectan a la salud.
¿Qué es la Epigenética?
La Epigenética estudia los cambios heredables y reversibles que ocurren en la función genética sin modificar la secuencia del ADN.
La salud física y mental no depende únicamente de las modificaciones en el código del ADN propias o heredadas de nuestros ancestros (genética), sino también de la interacción dinámica entre nuestros genes y el ambiente, aunque el código genético no se encuentre alterado (epigenética).
Los cambios epigenéticos son reversibles y dependen de la calidad de la interacción entre el individuo y su ambiente, y por lo tanto podemos actuar sobre ellos mejorando nuestra salud.

Influencia de la Epigenética:
Estudios en gemelos
Un ejemplo ilustrativo de la influencia de la epigenética se observa en estudios realizados en gemelos idénticos monozigóticos. A pesar de que los dos comparten una secuencia de ADN idéntica, ambos desarrollan una predisposición muy distinta a contraer cierto tipo de enfermedades.
Esto se explica, en buena parte, porque ambos han estado expuestos a estímulos muy distintos (dieta, vida social, aficiones, vicios, estrés, etc), y sus genomas, aunque idénticos, se expresan de manera diferente.
¿Qué importancia tiene la epigenética en medicina?
La epigenética juega un papel fundamental en el desarrollo de las enfermedades prevalentes (cardiovasculares, cáncer y cerebrales). Así, por ejemplo, mecanismos como memoria y aprendizaje, deterioro cognitivo asociado a la vejez o trastornos del comportamiento, están en gran parte regulados epigenéticamente.
Biomarcadores epigenéticos
EuroEspes está desarrollando una serie de biomarcadores epigenéticos que permiten tanto la detección de enfermedades como la monitorización de la respuesta a los distintos tratamientos suministrados al paciente:
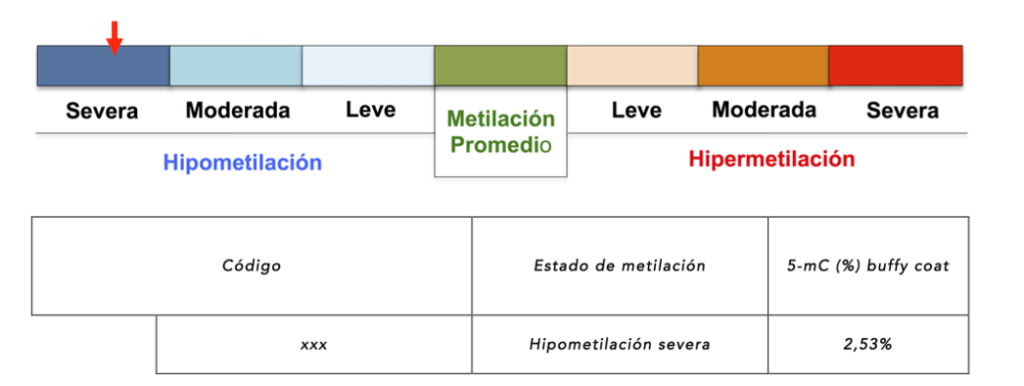
Metilación global del ADN
En procesos tumorales y neurodegenerativos existe unos niveles menores de metilación global del ADN. De este modo, este biomarcador permite detectar si esos niveles de metilación global corresponden a los de una persona sana, o si presenta unos niveles anómalos compatibles con procesos tumorales y neurodegenerativos. Además, el tratamiento con AtreMorine, un epinutraceútico que incrementa los niveles de Dopamina, aumenta los niveles de metilación global en pacientes con Parkinson.
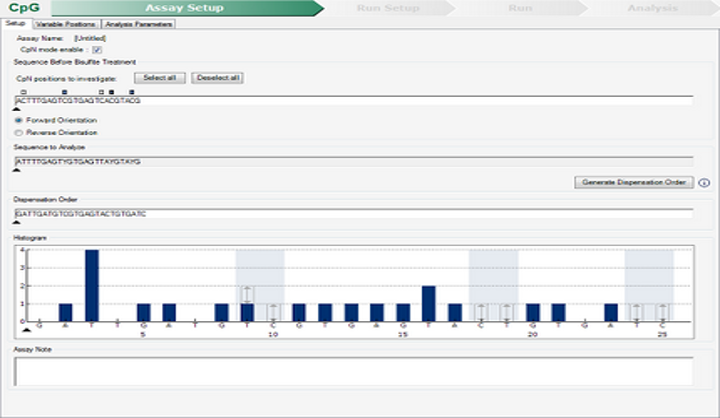
Pirosecuenciación
La técnica de pirosecuenciación permite la cuantificación de los niveles de metilación en determinadas regiones de algunos genes, permitiendo el diagnóstico de determinadas patologías (Parkinson, Alzheimer, cáncer colorrectal, etc.). Por ejemplo, la metilación de la Septina 9 está alterada en cáncer colorrectal. El estudio de los niveles de metilación de la septina 9 es un biomarcador para el cribado y diagnóstico precoz de este tipo de cáncer (aprobado por la FDA y la UE).
Expresión génica
Los cambios en los niveles de metilación provocan a menudo cambios en la expresión génica. De este modo, el estudio de los niveles de expresión de genes importantes en determinadas patologías supone también un nuevo biomarcador.
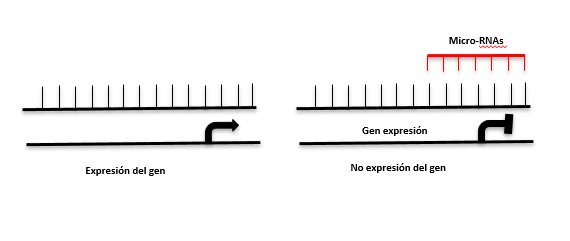
miRNAs
Los niveles de expresión de determinados miRNAs se asocian a distintas patologías. Por ejemplo, una firma epigenética de 12 miRNAs es un biomarcador para el diagnóstico precoz de cáncer de tiroides y otra firma de 8 miRNAs se emplea para el diagnóstico de cáncer de pulmón.
FarmacoEpigenética
Las diferencias entre individuos en la respuesta y metabolismo de los fármacos se explica tanto por la genética como por la epigenética de los individuos.
Se ha descrito, por ejemplo, que el fumar afecta a la metilación del gen CYP1A1, implicado en la metabolización de fármacos. Además, la metilación de distintos genes en procesos tumorales afecta a la respuesta a distintos agentes quimioterapéuticos. Por ejemplo, los niveles de metilación de MGMT están relacionados con la respuesta a la quimioterapia en pacientes con glioma.
De este modo, la combinación de estudios genéticos y epigenéticos supone un pilar fundamental de la medicina personalizada y permite un mejor tratamiento de los pacientes, evitando fármacos ineficaces o incluso tóxicos.
Entre los proyectos de I+D del Departamento de Epigenética Médica de EuroEspes, se encuentra el estudio de los cambios epigenéticos que ocurren en los genes implicados en metabolismo de fármacos y su asociación con la respuesta a dichos tratamientos.
Si tiene alguna duda o si necesita más información
no dude en ponerse en contacto con nosotros.

